 資訊處研究資訊組自102學年度(2013年8月)起開設「生醫資訊應用軟體」系列課程,選擇目前世界上常用且功能強大的分析軟體,定期邀請具多年使用經驗的專業團隊至校內講課,以提升一校三院各實驗室的資料分析能力。
資訊處研究資訊組自102學年度(2013年8月)起開設「生醫資訊應用軟體」系列課程,選擇目前世界上常用且功能強大的分析軟體,定期邀請具多年使用經驗的專業團隊至校內講課,以提升一校三院各實驗室的資料分析能力。
 至目前(2014年5月)為止已成功開辦:IPA整合分析軟體暨資料庫(Ingenuity Pathways Analysis)、基因體視覺化分析軟體(Integrative Genomics Viewer, IGV)、生物晶片與次世代定序資料分析平台(GeneSpring GX)、分生實驗軟體(DNASTAR Lasergene),與全基因組分析軟體(Golden Helix)等課程,其中約70%的學員為實驗室主持人或臨床醫師,可見一校三院對於資料分析的強烈需求。【圖:生醫資訊應用軟體系列課程上課實況】
至目前(2014年5月)為止已成功開辦:IPA整合分析軟體暨資料庫(Ingenuity Pathways Analysis)、基因體視覺化分析軟體(Integrative Genomics Viewer, IGV)、生物晶片與次世代定序資料分析平台(GeneSpring GX)、分生實驗軟體(DNASTAR Lasergene),與全基因組分析軟體(Golden Helix)等課程,其中約70%的學員為實驗室主持人或臨床醫師,可見一校三院對於資料分析的強烈需求。【圖:生醫資訊應用軟體系列課程上課實況】
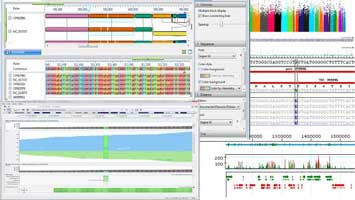
以次世代定序或生物晶片資料分析為例,整體分析步驟可分為基礎分析(primary analysis)、進階分析(downstream analysis)、統合分析(meta analysis)3個層次。
1.基礎分析:以實驗儀器產生的原始資料進行運算,產生如SNP variant call或heat map等簡單統計圖表,此步驟運用實驗儀器廠商所附帶提供的軟體即可完成,若為外包實驗,則外包廠商多半會免費幫忙完成基礎分析工作,故並非問題所在。
2.進階分析:使用基礎分析所產生的結果,依個別實驗之目的進行更深入之分析,例如:GWAS、haplotype analysis、differential expression analysis、alternative splicing analysis等,此部分若有強大的工具軟體協助,則各實驗室將有能力自行分析資料,可大幅提升研究產出效率。3.統合分析:通常需要對應實驗結果於外部資料庫,例如:gene set enrichment analysis(GSEA)等,並且與臨床資料或文獻資料交互驗證,因此需要串聯強大的整合資料庫工具,或是透過與專業分析團隊的合作來達成。【下圖:以工具軟體對人類基因體SNP進行GWAS分析】

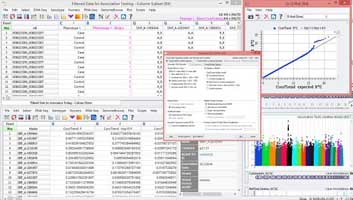
 目前研究資訊組的規劃以開設「進階分析」所需的軟體課程為主,以期能提升各實驗室本身的資料分析能力。在「統合分析」方面,亦將於103學年度採購IPA整合分析軟體暨資料庫的群體授權,提供給各實驗室及本校生物資訊核心設施等單位使用。若您對資料分析工具軟硬體有任何需求或建議,非常歡迎與研究資訊組聯繫,本組將會對一校三院進行需求調查,並依調查結果辦理開設訓練課程或統一採購等事宜。(文/張祐誠,資訊處研究資訊組組長、醫資所助理教授)【圖:約70%的學員為實驗室主持人或臨床醫師】
目前研究資訊組的規劃以開設「進階分析」所需的軟體課程為主,以期能提升各實驗室本身的資料分析能力。在「統合分析」方面,亦將於103學年度採購IPA整合分析軟體暨資料庫的群體授權,提供給各實驗室及本校生物資訊核心設施等單位使用。若您對資料分析工具軟硬體有任何需求或建議,非常歡迎與研究資訊組聯繫,本組將會對一校三院進行需求調查,並依調查結果辦理開設訓練課程或統一採購等事宜。(文/張祐誠,資訊處研究資訊組組長、醫資所助理教授)【圖:約70%的學員為實驗室主持人或臨床醫師】

