 臺北醫學大學為完善生物醫學整體研究,資訊處研究資訊組串連研發處共同儀器中心於2019年4月初建置代謝體資料分析平臺;透過共同儀器中心發展代謝產物測定流程,利用TOF-MASS(time-of-flight mass spectrometry),對樣本進行定性與定量分析,可獲得代謝體訊號。
臺北醫學大學為完善生物醫學整體研究,資訊處研究資訊組串連研發處共同儀器中心於2019年4月初建置代謝體資料分析平臺;透過共同儀器中心發展代謝產物測定流程,利用TOF-MASS(time-of-flight mass spectrometry),對樣本進行定性與定量分析,可獲得代謝體訊號。
近年來為更完整探索生物體運作機制,基因體學(Genomics)、轉錄體學(Transcriptomics)與蛋白質體學(Proteomics)已被廣泛利用於各研究中,隨著高通量生物技術之快速發展,代謝產物研究成為熱門的研究方向。代謝體學(Metabolomics)是研究小分子,例如:生物體內的基質與一級和二級代謝產物,而且這些小分子易受到基因與環境的變化而改變。因此,相較於其他體學,代謝體研究更能反應出環境變化對生物體的影響。目前代謝體學研究,多使用質譜儀進行定量與定性分析。在質譜儀產生資料後,須透過資料前處理(去除雜訊)、統計與圖像化表示等方式加以闡述,方能有良好的研究成果表現。
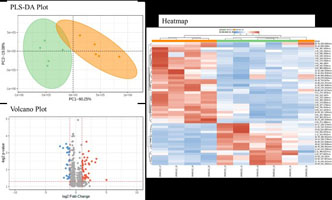 研究資訊組為此建置代謝體資料分析平臺,透過統計與圖像化表示,方便校內師生瞭解並闡述其研究之結果。例如,透過PLS-DA繪圖,可了解不同比較組別是否具有差異表現的代謝產物;並運用volcano plot與heatmap,可找尋有差異代謝產物;MASS Spectrum繪圖,可直接觀看不同比較組別中有差異代謝產物;運用交集或聯集方式,可找尋不同比較組別中,是否具有特定代謝產物,或多套相同實驗樣本中是否具有相似代謝產物。【右圖:PLS-DA(左上)、Volcano(左下)與Heatmap(右)的繪圖,可方便找尋不同群組中有差異帶謝產物】
研究資訊組為此建置代謝體資料分析平臺,透過統計與圖像化表示,方便校內師生瞭解並闡述其研究之結果。例如,透過PLS-DA繪圖,可了解不同比較組別是否具有差異表現的代謝產物;並運用volcano plot與heatmap,可找尋有差異代謝產物;MASS Spectrum繪圖,可直接觀看不同比較組別中有差異代謝產物;運用交集或聯集方式,可找尋不同比較組別中,是否具有特定代謝產物,或多套相同實驗樣本中是否具有相似代謝產物。【右圖:PLS-DA(左上)、Volcano(左下)與Heatmap(右)的繪圖,可方便找尋不同群組中有差異帶謝產物】
在表格當中,除了代謝產物預測外,亦提供HMDB (Human Metabolome Database)與KEGG (Kyoto Encyclopedia of Genes and Genomes)對應的化合物編號,可藉由HMDB資料庫編號,查找相關資訊,也可透過此分析平臺查詢KEGG pathway。此外,在分析平臺中,所有圖表皆可下載,並進行後續分析。例如,運用表格中的化合物列表,搭配生物途徑分析軟體IPA與MetaCore進行更進一步pathway enrichment analysis,此流程平臺之建立將有助於北醫大在代謝體研究領域之深入發展。(文/資訊處)【下圖:代謝體資料分析平臺具有資料管理、資料視覺化與表格下載等功能】


